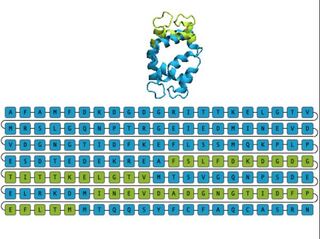
Microsoft представила универсальную структуру EvoDiff, которая, по утверждению компании, может генерировать «высокоточные» и «разнообразные» белковые последовательности. В отличие от других фреймворков EvoDiff не требует никакой структурной информации о целевом белке.
Старший научный сотрудник Microsoft Кевин Янг отметил, что EvoDiff можно применять для создания ферментов, которые откроют новые методы лечения и доставки лекарств, а также новых ферментов для промышленных химических реакций.
«Мы предполагаем, что EvoDiff расширит возможности белковой инженерии за пределы парадигмы структура-функция в сторону программируемого дизайна, ориентированного на последовательность», — отметил Ян.
Ядром платформы EvoDiff стала модель с 640 млн параметров, обученная на данных о различных видах и функциональных классах белков. Данные для обучения были получены из набора OpenFold для выравнивания последовательностей, а также UniRef50, базы данных последовательностей и функциональной информации, поддерживаемой консорциумом UniProt.
EvoDiff — это диффузионная модель, по архитектуре похожая Stable Diffusion и DALL-E 2. EvoDiff учится постепенно убирать шумы из исходного белка, с каждым шагом приближаясь к белковой последовательности.
Старший научный сотрудник Microsoft Ава Амини говорит, что EvoDiff может не только создавать новые белки, но и «заполнять пробелы» в существующей конструкции белка. При наличии части, которая связывается с другим белком, модель может генерировать аминокислотную последовательность, которая, например, соответствует набору критериев.
EvoDiff также может синтезировать «неупорядоченные белки». Они играют важную роль в развитии заболеваний, усиливая или снижая активность других белков.
Пока исследование EvoDiff не было рецензировано. Microsoft предстоит проделать «гораздо больше работы по масштабированию», прежде чем инфраструктуру можно будет использовать в коммерческих целях.
В качестве следующего шага команда EvoDiff планирует протестировать белки, созданные моделью, в лаборатории, чтобы определить, жизнеспособны ли они. Далее исследователи начнут работу над следующим поколением фреймворка.
В 2021 году компания DeepMind выпустила версию своей нейросети с глубоким обучением AlphaFold 2 для определения трёхмерной структуры белков. DeepMind показала, как её программное обеспечение может точно предсказывать структуру многих белков, используя только их последовательность, которая определяется ДНК. Исследователи работали над своей системой в течение десятилетий, и AlphaFold 2 отлично показала себя в рамках критической оценки прогнозирования структуры белка CASP, решив 50-летнюю проблему фолдинга или «сворачивания» белков.
В 2022 году исследователи Массачусетского технологического института создали модель машинного обучения, которая может предсказывать комплекс, образуемый при соединении двух белков. Их метод в 80–500 раз быстрее существующих, и часто предсказывает структуры белков, которые ближе к наблюдаемым экспериментально.
Источник новости: habr.com